دیپ مایند یکی از بزرگترین چالشهای زیست شناسی را حل میکند
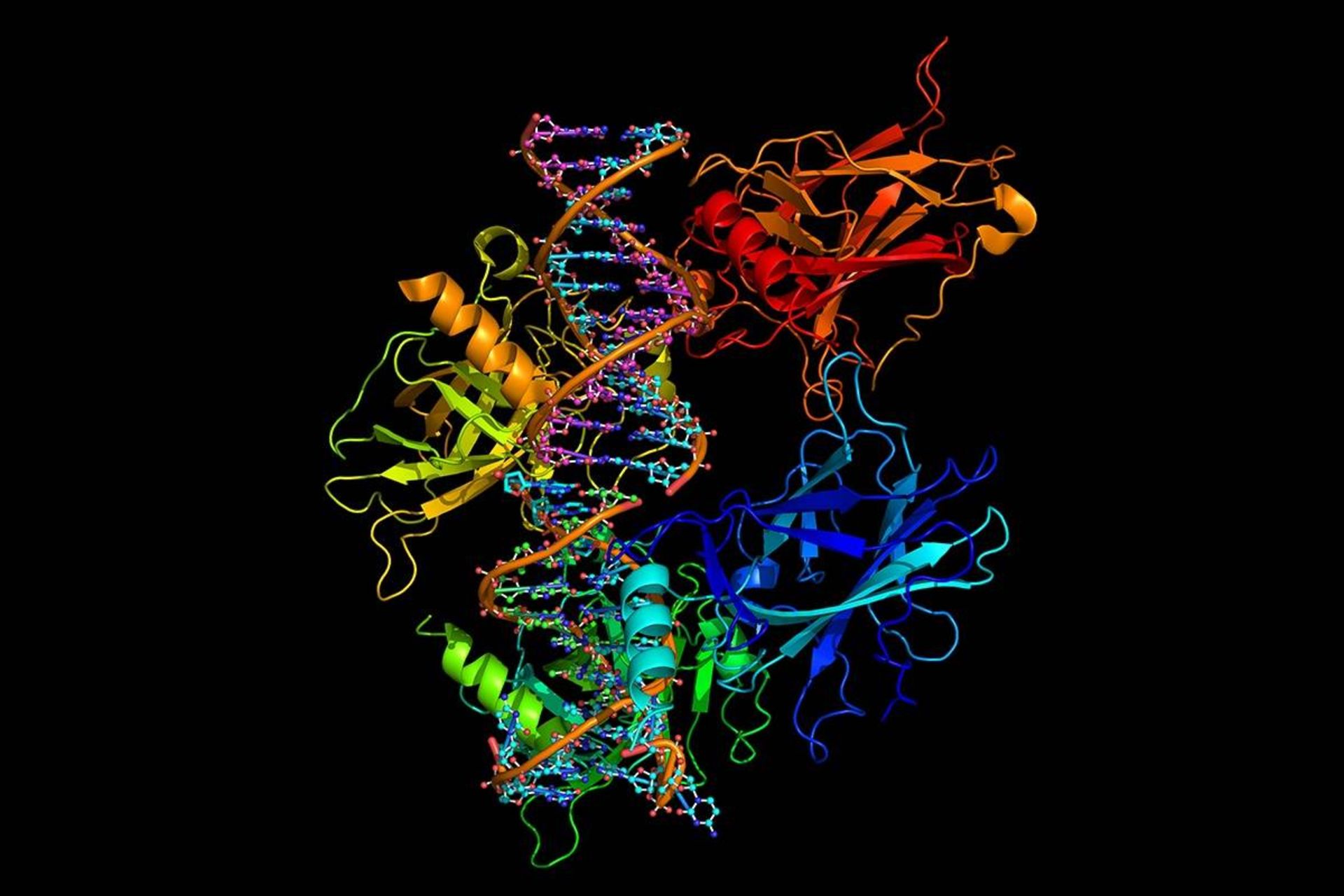
یک شبکهی هوش مصنوعی (AI) که توسط شاخهی دیپمایند (DeepMind) هوش مصنوعی گوگل ایجاد شده، به پیشرفت عظیمی در زمینهی حل یکی از بزرگترین چالشهای زیستشناسی یعنی تعیین شکل سهبعدی پروتئین براساس توالی آمینواسیدی آن دست پیدا کرده است. برنامهی دیپمایند که آلفافولد (AlphaFold) نام دارد، در چالش پیشبینی ساختار پروتئین دوسالانهای به نام CASP (ارزیابی انتقادی پیشگویی ساختار پروتئین) از حدود ۱۰۰ تیم دیگر بهتر عمل کرد. جان مولت، متخصص زیستشناسی محاسباتی در دانشگاه مریلند در کالج پارک، یکی از بنیانگذاران CASP در سال ۱۹۹۴ بود که با هدف بهبود روشهای محاسباتی برای پیشبینی دقیق ساختارهای پروتئین تأسیس شد. او میگوید: «این کار بسیار مهمی است. از یک جهت، میتوان گفت مسئله حل شده است.»
توانایی پیشبینی دقیق ساختارهای پروتئینی از روی توالی آمینواسیدی آنها مزیت بسیار بزرگی برای علوم زیستی و پزشکی است. این دستاورد تا حد زیادی تلاش برای درک واحدهای سازنده سلول را تسریع و کشف سریعتر و پیشرفتهتر داروها را ممکن خواهد کرد.
آلفافولد در CASP گذشته در سال ۲۰۱۸ که اولین مشارکت دیپمایند مستقر در لندن در این رقابت بود، در بالای جدول قرار گرفت. اما امسال این شبکهی یادگیری عمیق کاملا بالاتر از تیمهای دیگر بود و به گفتهی دانشمندان، عملکرد چنان خوبی داشت که میتواند قاصد انقلابی در زیستشناسی باشد. آندری لوپاس، متخصص زیستشناسی تکاملی از مؤسسه زیستشناسی تکوینی ماکس پلانک در آلمان که عملکرد تیمهای مختلف را در CASP ارزیابی کرد، آن را تحولآفرین خواند. آلفافولد قبلا به لوپاس کمک کرده است ساختار پروتئینی را پیدا کند که به مدت یک دهه آزمایشگاه او را سردرگم کرده بود و وی پیشبینی میکند این پیشرفت باعث تغییر نحوهی کار و سؤالهایی شود که با آن برخورد میکند.
در برخی موارد، پیشبینیهای آلفافولد از موارد حاصل از روشهای تجربی که بهعنوان استاندارد طلایی درنظر گرفته میشود، قابل تمایز نبود: مانند کریستالوگرافی اشعه ایکس یا پراش اشعه ایکس و در سالهای اخیر، کرایو میکروسکوپی الکترونی (cryo-EM). دانشمندان میگویند آلفافولد ممکن است نیاز به این روشهای سخت و گرانقیمت را برطرف نکند؛ اما هوش مصنوعی امکان مطالعهی موجودات زنده را به روشهای جدید فراهم میکند.
مسئله ساختار پروتئین
پروتئین بهعنوان واحد سازنده حیات، مسئول بیشتر اتفاقاتی است که درون سلول رخ میدهد. نحوهی عمل پروتئین و کاری که انجام میدهد، بهوسیلهی شکل سهبعدی آن تعیین میشود. پروتئینها معمولا بدون کمک و فقط براساس قوانین فیزیک شکل خود را به دست میآورند.
برای چندین دهه، متدهای آزمایشگاهی اصلیترین روش برای دستیابی به ساختارهای پروتئینی بوده است. اولین ساختارهای کامل از پروتئینها در دههی ۱۹۵۰ با استفاده از تکنیکی به دست آمد که در آن پرتوهای ایکس به پروتئینهای کریستالشده شلیک و نور تفرقیافته به مختصات اتمی پروتئین ترجمه میشود. کریستالوگرافی اشعه ایکس سهم زیادی در تعیین ساختارهای پروتئینی داشته است. اما طی دههی گذشته، cryo-EM به ابزار مورد علاقهی بسیاری از آزمایشگاههای فعال درزمینهی زیستشناسی ساختاری تبدیل شده است.
مدتها دانشمندان در این مورد کنجکاو بودهاند که چگونه اجزای تشکیلدهندهی پروتئین (رشتهای از اسیدهای آمینهی مختلف) پیچوتابهای شکل نهایی پروتئین را طرحریزی میکند. تلاشهای اولیه برای استفاده از کامپیوتر برای پیشبینی ساختارهای پروتئین در دهههای ۱۹۸۰ و ۱۹۹۰ عملکرد ضعیفی داشته است و روشهایی که در مقالات در مورد آنها ادعاهای زیادی میشد، وقتی توسط دانشمندان دیگر روی پروتئینهای متفاوت آزمایش میشد، حاصلی نداشت.
مولت CASP را باهدف سختگیری بیشتر در مورد این تلاشها راهاندازی کرد. این رویداد تیمها را به چالش میکشد تا ساختارهای پروتئینهایی را پیشبینی کنند که با استفاده از روشهای تجربی حل شده است؛ اما ساختار آنها هنوز دردسترس قرار نگرفته است.
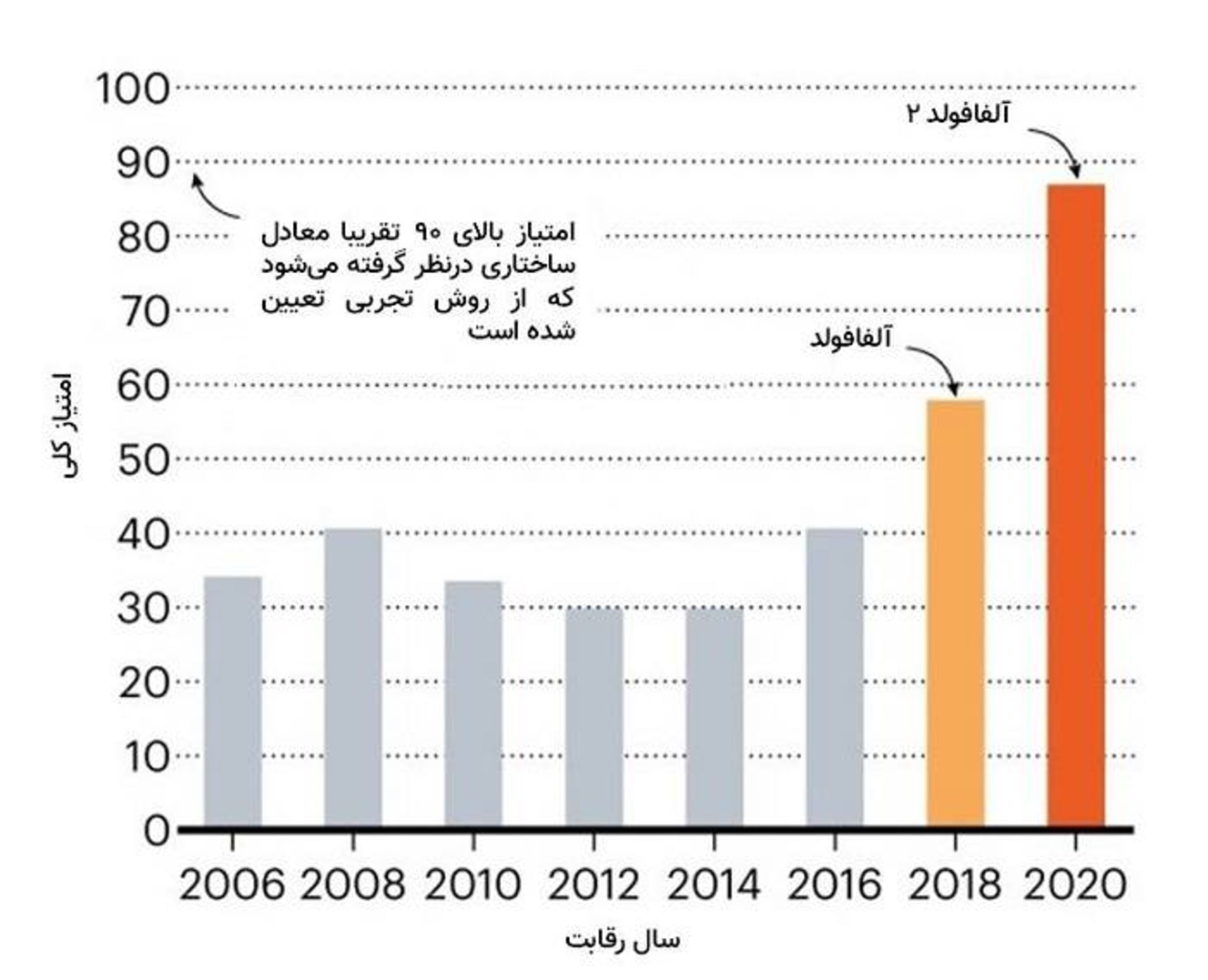
الگوریتم آلفافولد ۲ دیپمایند در رقابت CASP14 در زمینهی پیشبینی ساختار سهبعدی پروتئین بهطور قابلتوجهی نسبت به دیگر تیمها بهتر عمل کرد؛ نسخهی قبلی آن (آلفافولد) نیز در رقابت CASP قبلی نسبتبه گروههای دیگر عملکرد بهتری داشت
عملکرد سال ۲۰۱۸ دیپمایند در CASP13 موجب تحیر بسیاری از دانشمندان شد. اولین تکرار آلفافولد، روشی از هوش مصنوعی که با عنوان یادگیری عمیق شناخته میشود، در مورد دادههای ژنتیکی و ساختاری به کار میبرد تا فاصلهی جفتهای اسید آمینه در یک پروتئین را پیشبینی کند. جان جامپر که هدایت پروژه را برعهده داشته است، میگوید آلفافولد در دومین مرحله که در آن هوش مصنوعی را فراخوانی نمیکند، از این اطلاعات برای تهیهی یک مدل اجماع (consensus model) درمورد پیشبینی شکل پروتئین استفاده میکند. تیم سعی داشت این رویکرد را دنبال کند؛ اما درنهایت به بنبست رسید. بنابراین روند را کاملا تغییر داد و شبکهی هوش مصنوعی را ایجاد کرد که شامل اطلاعاتی در مورد محدودیتهای فیزیکی و هندسی مرتبط با نحوهی تاخوردن پروتئینها نیز میشد. آنها همچنین وظیفهی دشوارتری را برای آن تعیین کردند: شبکه به جای پیشبینی روابط اسیدهای آمینه، ساختار نهایی توالی پروتئین هدف را پیشبینی میکند.
دقت حیرتانگیز
رقابت CASP در طول چند ماه برگزار میشود. پروتئینهای هدف یا بخشهایی از پروتئینها که دومِین نامیده میشود (درمجموع حدود ۱۰۰ مورد)، با فواصل منظمی منتشر میشوند و تیمها چند هفته فرصت دارند تا پیشبینیهای ساختاری خود را ارائه دهند. سپس گروه مستقلی از دانشمندان پیشبینیها را با استفاده از معیارهایی ارزیابی میکنند و مشخص میکنند که پروتئین پیشبینیشده تا چه حد با ساختاری که به شکل تجربی تعیین شده است، تشابه دارد. ارزیابان نمیدانند چه گروههایی این پیشبینیها را انجام دادهاند.
پیشبینیهای آلفافولد تحت نام «گروه ۴۲۷» وارد رقابت شد. برخی پیشبینیها بهتر از دیگران بود؛ اما تقریبا دو سوم از پیشبینیها از نظر کیفیت با ساختارهای تجربی قابل مقایسه بود. مولت میگوید در برخی موارد، مشخص نبود آیا اختلاف پیشبینیهای آلفافولد و نتایج تجربی، خطای پیشبینی است یا حاصل خطای روش تجربی.
پیشبینیهای آلفافولد با ساختارهای تجربی تعیینشده بهوسیلهی تکنیکی به نام تصویربرداری تشدید مغناطیسی هستهای همخوانی ضعیفی داشت که به گفتهی مولت، میتواند ناشی از نحوهی تبدیل دادههای خام به مدل باشد.
این شبکه همچنین در زمینهی مدلسازی ساختارهای جداگانه به کمپلکسها یا گروههای پروتئینی به مشکل برمیخورد که به موجب آن، تعامل با پروتئینهای دیگر اشکال آنها را تغییر میدهد. مولت میگوید بهطور کلی، امسال در مقایسه با CASP گذشته، پیشبینیهای گروههای مختلف در مورد ساختار پروتئینها دقیقتر بود؛ اما بیشترین پیشرفت را میتوان مربوط به آلفافولد دانست.
به گفتهی مولت، در مورد اهداف پروتئینی دارای دشواری متوسط، بهترین عملکرد تیمهای دیگر معمولا از مقیاس دقت ۱۰۰ امتیازی، ۷۵ امتیاز کسب میکرد، درحالیکه روی همین اهداف، امتیاز آلفافولد حدود ۹۰ بود. مولت میگوید تقریبا نصف تیمها در خلاصهی رویکرد خود، یادگیری عمیق را ذکر کرده بودند که نشان میدهد هوش مصنوعی تأثیر گستردهای در این زمینه از علم خواهد داشت. بیشتر شرکتکنندگان گروههای دانشگاهی بودند؛ اما مایکروسافت و شرکت فناوری چینی تنسنت نیز وارد CASP14 شدند.
محمد القریشی، متخصص زیستشناسی محاسباتی از دانشگاه کلمبیا در شهر نیویورک و یکی از شرکتکنندگان CASP، مشتاق است که جزئیات عملکرد آلفافولد در این رقابت را مورد بررسی قرار دهد و درمورد نحوهی کار این سیستم اطلاعات بیشتری کسب کند. او میگوید احتمال بسیار کمی وجود دارد که علت عملکرد بالای این رویکرد، ساده بودن اهداف پروتئینی باشد؛ اما وی بیشتر بر این باور است که آلفافولد تحولآفرین خواهد بود. او میگوید: «فکر میکنم منصفانه است که بگوییم آلفافولد در زمینهی پبیشبینی ساختار پروتئین تحولآفرین خواهد بود. حدس میزنم افراد زیادی این زمینه را ترک کنند؛ زیرا به احتمال زیاد مسئلهی اصلی حل شده است. »
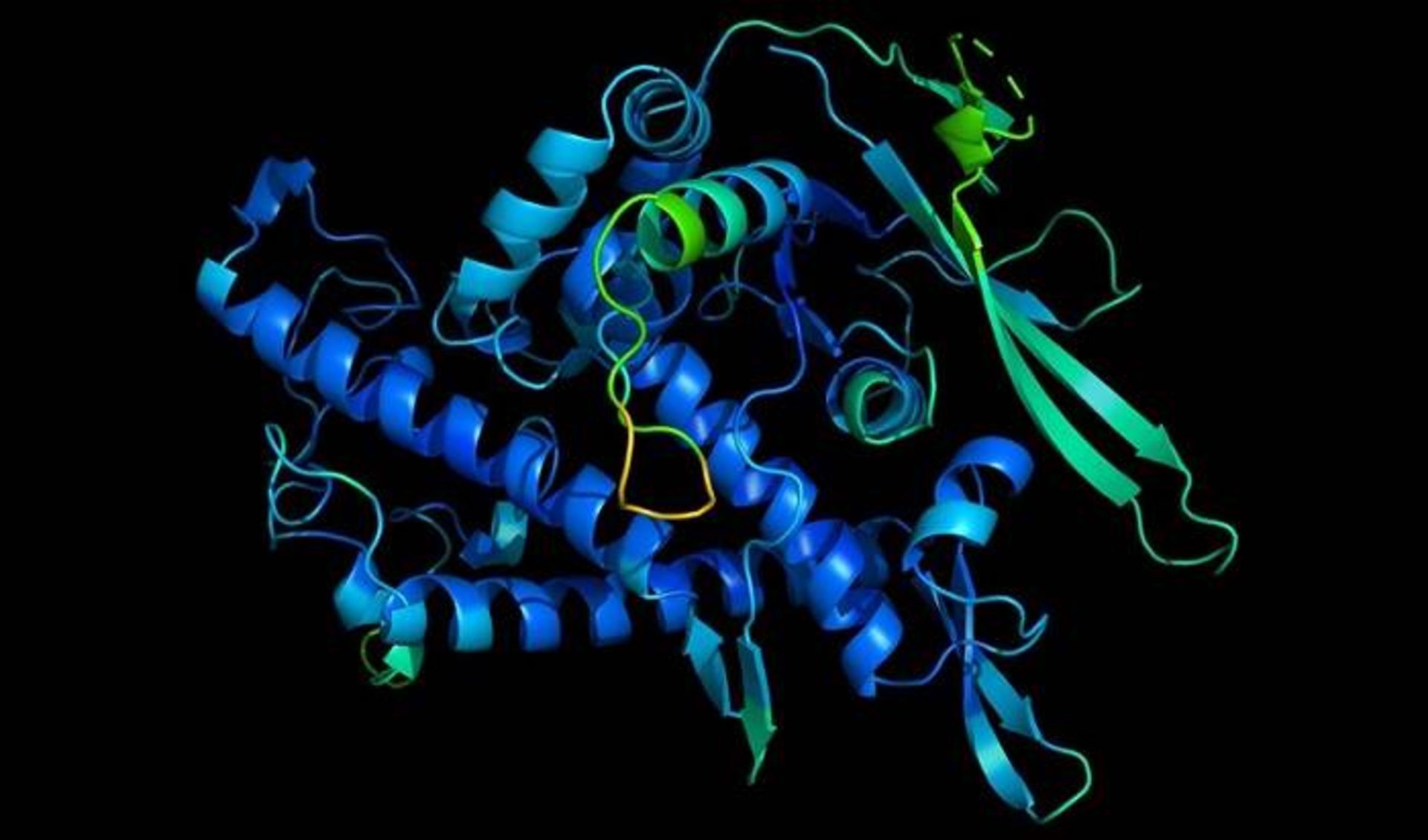
عملکرد پروتئین بهوسیلهی شکل سهبعدی آن تعیین میشود
ساختارهای سریعتر
یکی از پیشبینیهای آلفافولد به تعیین ساختار نوعی پروتئین باکتریایی کمک کرد که آزمایشگاه لوپاس سالها در حال تلاش برای کشف آن بود. تیم لوپاس پیش از این دادههای خام پراش اشعه ایکس را جمعآوری کرده بود؛ اما تبدیل این الگوهای لکهمانند به ساختار، نیازمند قدری اطلاعات در مورد شکل پروتئین است. ترفندهایی که برای به دست آوردن این اطلاعات استفاده شد و نیز ابزارهای پیشبینی دیگر با شکست روبهرو شدند. لوپاس میگوید: «پس از اینکه یک دهه صرف آزمایش هر چیزی کرده بودیم، مدل گروه ۴۲۷ ظرف نیم ساعت ساختار را به ما داد.»
دمیس هاسابیس، همبنیانگذار و مدیر اجرایی دیپمایند، میگوید این شرکت قصد دارد آلفافولد را بهمنظور استفادهی دانشمندان دیگر اصلاح کند. ممکن است چند روز طول بکشد تا آلفافولد ساختار پیشبینیشدهای ارائه دهد که شامل برآوردهای مربوط به قابلیت اعتماد مناطق مختلف پروتئین میشود. هاسابیس که کشف دارو و طراحی پروتئین را از کاربردهای بالقوه روش خود میداند، میافزاید: «ما تازه شروع به درک چیزی کردهایم که زیستشناسان بهدنبال آن هستند.»
در اوایل سال ۲۰۲۰، این شرکت پیشبینیهایی از ساختارهای چند پروتئین SARS-CoV-2 منتشر کرد که هنوز با استفاده از روش تجربی تعیین نشده بود. استفان بروهاون، متخصص عصبزیستشناسی مولکولی از دانشگاه کالیفرنیا که تیمش ساختارهای مذکور را در ماه ژوئن منتشر کرد، میگوید پیشبینیهای دیپمایند در مورد پروتئینی بهنام Orf3a، بسیار شبیه چیزی بود که بعدا با استفاده از cryo-EM تعیین شد.
تأثیر در دنیای واقعی
بعید است که آلفافولد موجب توقف کار آزمایشگاههایی مانند آزمایشگاه بروهاون شود که از روشهای تجربی برای حل ساختارهای پروتئینی استفاده میکند. اما این دستاورد میتواند به معنای آن باشد که دادههای تجربی دارای کیفیت پایینتر و از نظر جمعآوری سادهتر، تمام چیزی خواهند بود که برای رسیدن به ساختاری مناسب لازم هستند.
برخی کاربردها نظیر تجزیهوتحلیل تکاملی از پروتئینها ممکن است رونق بگیرد؛ زیرا حجم عظیمی از دادههای ژنومی دردسترس است که ممکن است بتواند بهطور قابل اعتمادی به ساختار تبدیل شود. جانت تورنتون، متخصص زیستشناسی ساختاری و یکی از ارزیابهای CASP گذشته، میگوید: «فکر میکردم این مسئله در طول زندگی من حل نخواهد شد.» او امیدوار است این روش بتواند به آشکار شدن عملکرد هزاران پروتئین حلنشده در ژنوم انسان کمک کند و تغییرات ژنی عامل بیماری را که در افراد متفاوت است، معنا کند.
عملکرد آلفافولد نقطهی عطفی برای دیپمایند محسوب میشود. این شرکت به خاطر دارا بودن هوش مصنوعی مسلط بر بازیهایی مانند گو مشهور است؛ اما هدف بلندمدت آن توسعهی برنامههایی بوده است که بتواند به هوش کلی نظیر هوش انسان دست پیدا کند. هاسابیس میگوید حل چالشهای علمی بزرگ مانند پیشبینی ساختار پروتئین، یکی از مهمترین کاربردهایی هوش مصنوعی آنها است.
نظرات